蛋白質對于人體和生命來說
猶如高樓大廈中的一磚一瓦
失去蛋白質,生命也會如同
缺少材料的大樓般轟然倒塌
人體中存在眾多類型的蛋白質,如人體免疫系統的抗體蛋白、膠原蛋白、抗凍蛋白、核糖體等,每種蛋白質都有自己獨特的蛋白質三維結構。研究蛋白質,對于生命科學與藥物研發具有重要的價值。
很多人體疫病,都是由蛋白質的錯誤折疊引發的,比如帕金森癥、阿爾斯海默癥、亨廷頓癥等。藥物原理類似于一把鑰匙,鎖就是疫病靶點,通常可以把它認為是一種蛋白質。“鑰匙小分子”加入鎖孔就是和蛋白質發生結合,抑制蛋白質正常作用,或者激活蛋白質的某些作用。
因此,了解和預測蛋白質的形狀,有利于科學家設計出新的更有效的治療疾病的方法,幫助新藥物發現,降低實驗成本。
蛋白質是由氨基酸序列組成,但真正決定蛋白質作用的是它的3D結構,即氨基酸序列的折疊方式,如下圖的示例。蛋白質結構發現的主要方法,包括X-ray晶體衍射法、核磁共振法,以及2013年后成為熱門的冷凍電鏡三維重構法等。但是冷凍電鏡采購成本高昂,圖像重構需要耗費大量的計算力,往往需要很長時間才能解出一個新的蛋白質3D結構。
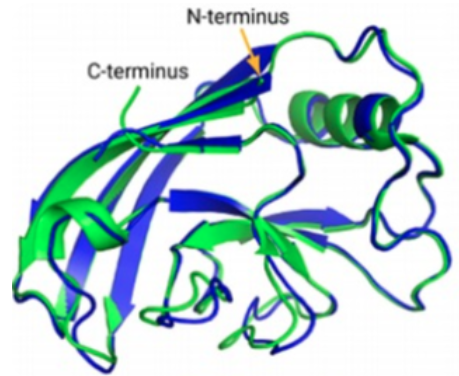
1972年諾貝爾化學獎得主,美國生物化學家克里斯蒂安. 安芬森提出:給定一個氨基酸序列,理論上就能預測出蛋白質的3D結構。五十年來,為了驗證這個理論,科學家嘗試各種模型預測蛋白質結構的方法,但是在全球蛋白質結構預測領域最知名的CASP競賽中,直到2018年預測準確率的成績只是40%上下。
蛋白質越大,模型越復雜和困難,因為需要考慮氨基酸之間更多相互作用。據統計,枚舉一個蛋白質可能的構型平均有10的300次冪的搜索空間。采用傳統的如分子動力學結構預測計算方案,需要極高的算力以及漫長的計算時間。在過去50年的時間內,只有17%的人類蛋白質組得到結構解析。

2020年Google DeepMind推出的AlphaFold2改變了一切。2020年12月發布的CASP14成績單,AlphaFold2將CASP蛋白質結構預測成績提高到92.4分(滿分100分),與蛋白質真實結構只差一個原子的寬度。2021年7月,Alphafold2模型結構及訓練過程發布在Nature雜志,并開源了蛋白質結構數據庫及推理代碼。
Alphafold2能夠預測出98.5%的人類蛋白質結構,其中60%的結構位置預測具有可信度。Science雜志則把AlphaFold2評選為2021年十大科學發現之首。
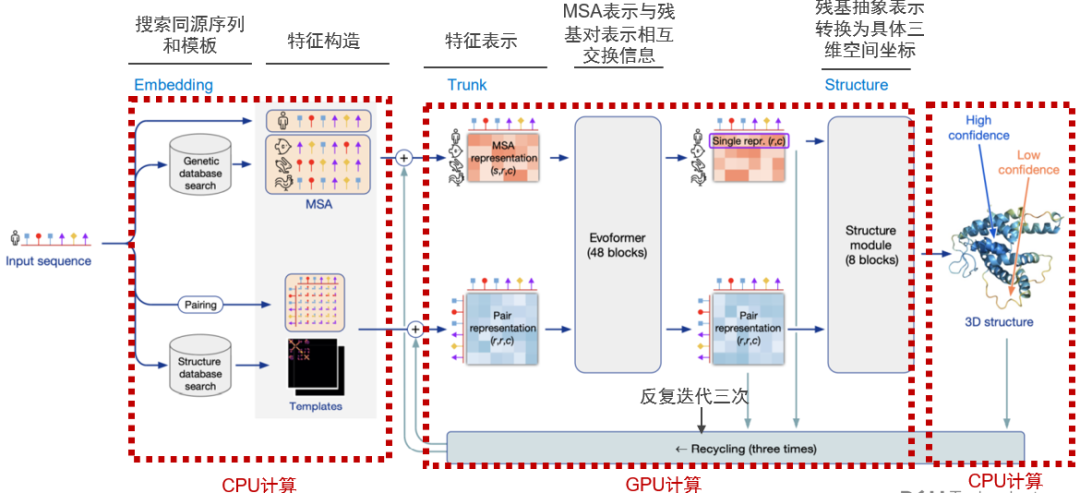
Alphafold2相較第一代AlphaFold的卷積神經網絡,利用多序列比對(MSA),將蛋白質的結構和生物信息整合到深度學習算法,主要包括神經網絡EvoFormer和結構模塊Structure Module:
EvoFormer主要將圖網絡和多序列比對結合完成結構預測,圖網絡將蛋白質相關信息構建成一個圖表,以此表示不同氨基酸質間的距離;通過三重Attention自注意力機制來處理氨基酸之間的關系圖。結構模塊主要將EvoFormer得到的信息轉換為蛋白質的3D結構。AlphaFold2是一個端到端神經網絡,反復將最終損失應用于輸出結果,然后對輸出結果進行遞歸,以不斷逼近正確結果。
那么,訓練AlphaFold2以及使用AlphaFold2進行蛋白質結構預測的推理計算,需要怎樣的計算力支持?戴爾科技中國研究院以及戴爾數據中心業務部解決方案團隊,通過在GitHub下載AlphaFold2模型代碼,部署在Dell PowerEdge XE8545服務器上,使用NVIDIA A100 GPU測試AlphaFlod2對68-2750個氨基酸殘基組成的不同大小的蛋白質進行3D結構預測,對AlphaFold2的計算性能和特性進行評估。

戴爾PowerEdge XE8545是戴爾科技最新推出的15G服務器家族中,專門針對AI GPU計算進行設計和優化的加速服務器。4U空間內可以支持4張A100 GPU加速卡,GPU之間通過NVLink實現600GB/s的pear-to-pear高速直連通信。
測試環境硬件及軟件配置如下:
●AMD EPYC 7713 64-Core Processor × 2
●1024 GB memory
●Nvidia A100 GPUs × 4, 80GB/500W
●CentOS Linux 7.0
●Python 3.8.0, TensorFlow 2.5.0
●CUDA 11.5, cuDNN 8.3
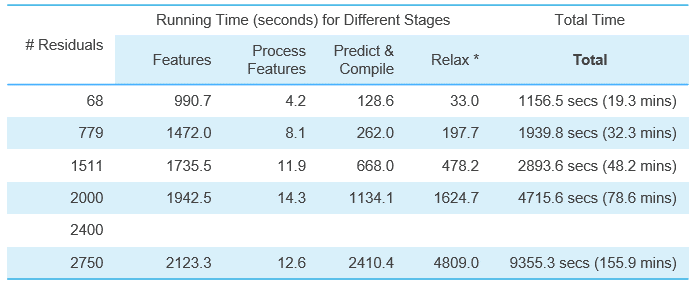
XE8545推理68-2750個氨基酸殘基組成的蛋白質的3D結構預測耗費的計算時間如下表所示(Top1模型,即推薦置信度最佳的模型),使用單張A100推理計算時間從19.3分鐘到2個半小時不等。
如果按照DeepMind論文Top5模型,XE8545單卡A100推理計算時間如下:
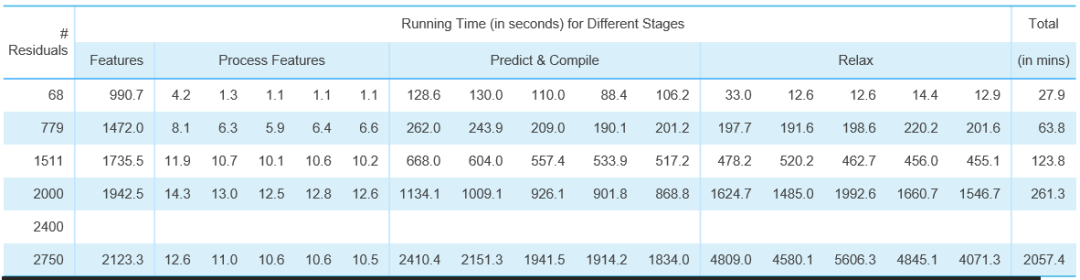
通過性能日志分析,我們可以明顯地看到AlphaFold2在推理過程中,由CPU和GPU交替計算,第一階段同源序列搜索、模版搜索及特征構造,以及最后階段3D結構生成的計算過程主要由CPU計算;中間第二個階段Evoformer神經網絡和結構模塊計算則主要由GPU進行計算。而XE8545所提供的強勁GPU算力與AMD 多核CPU算力(128核),則能夠確保AlphaFold2在規定時間內完成一個大型的蛋白質3D結構的預測計算。
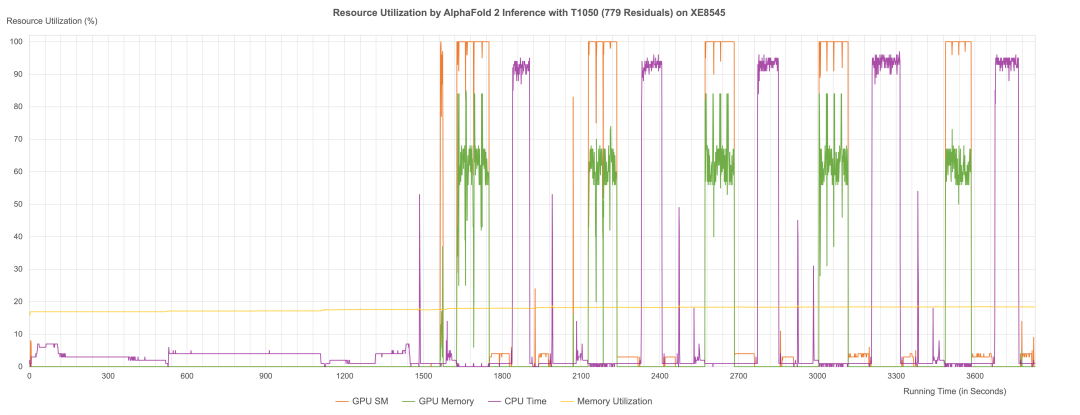
我們也對比了不同GPU對于AlphaFold2推理計算性能的影響。我們選取了一臺戴爾7750工作站,配置一張NVIDIA RTX5000顯卡,對蛋白質結構預測(Top1模型)計算性能進行對比,對比結果如下表所示:
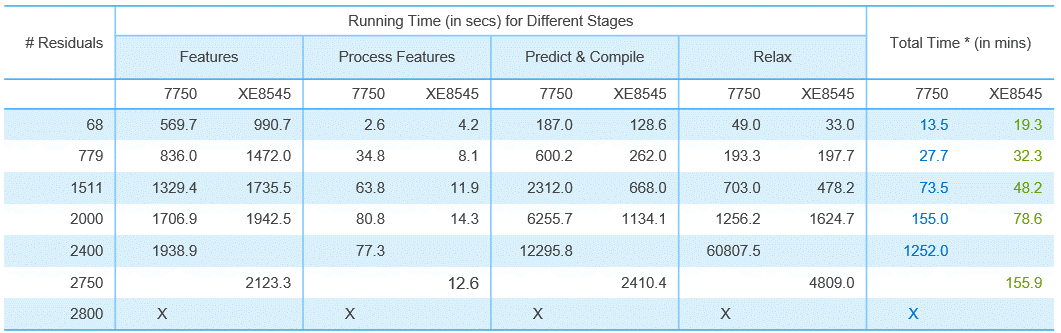
實驗數據顯示:當蛋白質規模很小的時候,企業級與消費級GPU性能相差不大;越大的蛋白質,使用A100結構預測加速性能越明顯。預測1511個殘基的蛋白質3D結構,XE8545+A100耗費時間是RTX5000的65%;預測2000個殘基的蛋白質3D結構,XE8545耗費的時間只有RTX5000的50%。
我們可以看到,當預測2800個殘基的蛋白質結構時,RTX5000由于顯存容量和算力的限制,無法完成結構預測工作,而XE8545仍然以小時級的時間順利完成同等規模的蛋白質結構預測。
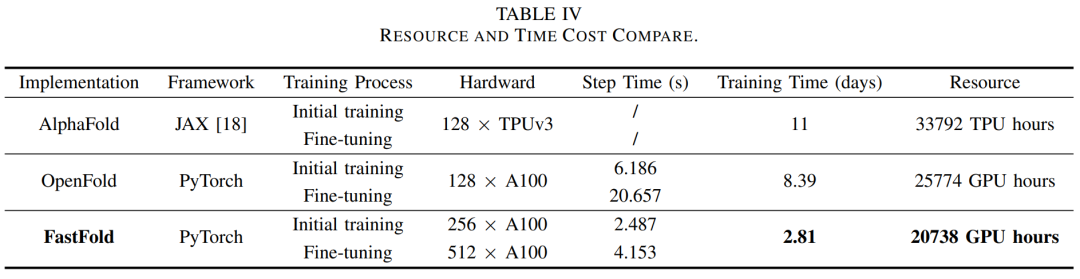
從模型訓練的角度來看,Alphafold2以及后續出現的類似的蛋白質結構預測模型,由于采用Transformer機制,模型訓練需要非常高的計算力,通常需要64-512張GPU組成計算集群,采用分布式訓練機制,才能在比較短的時間內實現模型收斂。
DeepMind在論文中談到,訓練AlphaFold2模型使用128塊Google TPU芯片,接近2周時間完成模型訓練。2022年3月,上海交通大學與潞晨科技發布的FastFold模型,使用256張A100 GPU進行初始訓練和512張A100進行Fine-tuning,2.81天完成模型訓練。
戴爾科技AI GPU分布式訓練解決方案,能夠提供高速GPU計算、小文件IO快速讀寫(蛋白質數據庫存在大量小文件)和高帶寬低延遲地網絡通信,幫助用戶實現在深度學習框架下分布式訓練的自動化實現與性能優化,輕松應對AI時代浪潮。
除此以外,2021年發布的《戴爾科技AI GPU分布式訓練技術白皮書》,還可以為用戶AI大模型GPU分布式訓練提供基礎架構解決方案、參考架構及優化建議。公眾號后臺回復關鍵字“白皮書”即可輕松獲取哦~


































































































 京公網安備 11010502049343號
京公網安備 11010502049343號